Seja na identificação do estágio dos casos da doença de Parkinson, ou para selecionar os melhores embriões para serem usados em um procedimento de fertilização, cada vez mais pesquisas têm explorado o potencial das tecnologias de inteligência artificial em processos de diagnósticos clínicos. Em uma nova investigação, liderada pelo docente da Unesp Leandro Alves Neves, do Departamento de Ciências de Computação e Estatística, câmpus de São José do Rio Preto, um grupo de pesquisadores buscou determinar a melhor combinação de técnicas para o diagnóstico e a classificação do chamado linfoma não-Hodgkin.
Os linfomas não-Hodgkin (LNH) são tumores malignos que afetam o sistema linfático, um componente do sistema imunológico, que é o responsável por auxiliar no combate a doenças e infecções. Segundo dados do Ministério da Saúde, o número de casos desse tipo de câncer duplicou nos últimos 25 anos. Dentre os 90 mil casos de câncer identificados entre 2010 e 2020, 74% foram classificados como linfoma não-Hodgkin. Segundo o Instituto Nacional de Câncer, apenas em 2022 foram diagnosticados mais de 12 mil novos casos.
Não é fácil detectar o LNH, pois ele apresenta sintomas comuns e que podem facilmente ser associados a outras doenças, como febre, suores noturnos, perda de peso, coceiras na pele e, o mais diferenciado, aumento de linfonodos do pescoço, axilas ou virilhas. Além disso, a patologia se caracteriza por grande diversidade, contabilizando-se nada menos do que vinte tipos diferentes de LNH. A essa diversidade corresponde uma variação nas células do sistema linfático que são atingidas pela patologia, o que, por sua vez, demanda tratamentos distintos. E a chave para encontrar o tratamento adequado, com as ações mais apropriadas para cada tipo de LNH, está no diagnóstico correto.
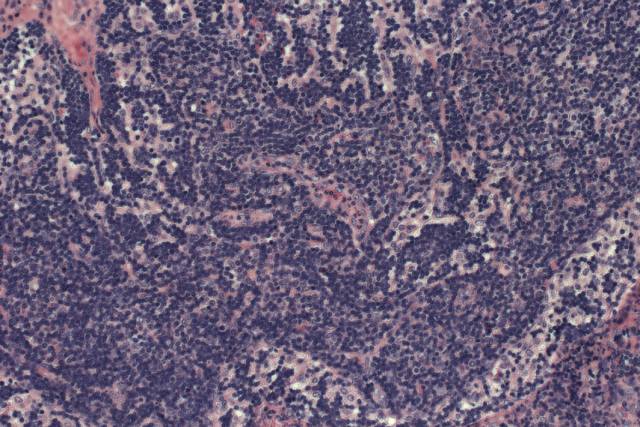
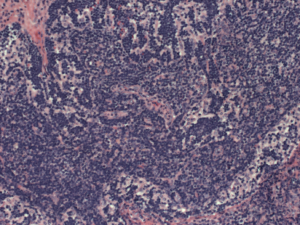
Atualmente, essa identificação se dá por meio de biópsia. Retiram-se amostras do tumor que a seguir passam por um processo de tingimento com corantes, como a hematoxilina e eosina (H&E). A etapa posterior envolve a condução de observações microscópicas por especialistas, em busca de sinais característicos do linfoma. Essa análise é extremamente complexa pois, além das dificuldades e subjetividades inerentes à classificação visual, há também a possibilidade de que um mesmo tumor venha a apresentar diferentes características em termos de tamanho, forma e padrão. O conjunto desses fatores pode dificultar a classificação correta, e isso tem estimulado pesquisadores a desenvolverem sistemas computacionais que possam dar suporte ao diagnóstico, auxiliando especialistas no processo de análise das amostras.
Focada em três tipos de LNH – Leucemia Linfoide Crônica, no Linfoma Folicular e no Linfoma de Células do Manto -, a pesquisa buscou formas de combinar técnicas computacionais e de preparação das imagens para encontrar a melhor combinação de métodos, de forma a resultar num crescimento do grau de precisão, tanto para o diagnóstico de linfoma não-Hodgkin, como para a classificação do tipo de LNH em questão.
Nesse novo método, os pesquisadores empregaram uma técnica que até então não havia sido aplicada para o estudo desse tipo de tumor, chamada de entropia amostral (SampEn).
O artigo, intitulado Classification of non-Hodgkin lymphomas based on sample entropy signatures, foi publicado na revista científica Expert Systems with Applications, e fez parte da pesquisa de mestrado de Guilherme Botazzo Rozendo, no programa de Pós-Graduação em Ciências da Computação da Unesp, sob orientação de Neves. A colaboração entre professor e estudante teve início desde cedo. “Antes de entrar na universidade eu trabalhei em um hospital como técnico de informática e gostei muito dessa área. Logo que comecei a graduação procurei um docente que atuasse na área com isso, que no caso era o professor Leandro”, lembra Rozendo. A dupla trabalha em conjunto desde 2014, primeiro ano da graduação de Rozendo, e a colaboração segue até hoje, com Neves como orientador de doutorado do jovem pesquisador.
A desordem pode ser um indicador
Concebido na física, o conceito de entropia serve para medir o grau de desordem das partículas em um sistema físico, ou seja, a quantidade de maneiras pelas quais as partículas conseguem gerar diferentes configurações. Ao transporem o conceito para a técnica computacional, os pesquisadores buscaram preservar o mesmo princípio: perceber o grau de desordem na imagem da amostra que está sendo analisada, e expressar esse grau de desordem de uma forma numérica. “Buscamos transformar aquilo que é originalmente visual em uma análise quantitativa, com valores que poderiam ser fornecidos para algoritmos de classificação”, diz Neves.
A técnica utiliza imagens capturadas por exames, e busca por padrões nelas. O sistema computacional seleciona aleatoriamente regiões dessa imagem, que são designadas de “janelas”, e compara essas janelas com outras partes da imagem, para determinar o grau de semelhança.
O fator de comparação é a textura da imagem. É a textura que vai informar ao sistema o grau de irregularidade que aquela “janela” apresenta. Quanto maior a discrepância da textura entre as regiões comparadas, maior é o valor de entropia. E quanto mais entropia houver naquela imagem, maior será a possibilidade da existência do linfoma. Por outro lado, se duas regiões sorteadas são muito semelhantes, a entropia é baixa, o que significa que a amostra não apresenta o câncer. Alterando o tamanho das janelas, ou do grau de tolerância para as irregularidades, os pesquisadores criaram assinaturas para cada imagem, representando a descrição numérica do comportamento observado na amostra sob análise. Essa descrição numérica serve para alimentar os algoritmos que, no final do processo, serão responsáveis por classificar os linfomas.
Colorir e dividir
Além da criação de assinaturas, os pesquisadores também avaliaram o uso da análise da entropia amostral em imagens que passaram por técnicas de tratamento comuns na prática clínica. “Uma vez que as imagens costumam ser tingidas com corantes H&E, é comum aplicar técnicas computacionais para tentar deixar essas imagens normalizadas, ou seja, minimizar a presença desse corante, que poderia afetar uma análise mais apropriada da imagem”, diz. O corante H&E, apesar de necessário para permitir que a amostra seja analisada ao microscópico, pode impactar a etapa de observação por um especialista, a depender da maneira como foi tingida a amostra e da concentração do corante. Por conta disso, após o tingimento as amostras têm suas cores e tons padronizados, a fim de permitir um estudo mais preciso.
Nesse sentido, o grupo também investigou o uso da técnica de entropia amostral em imagens tingidas a partir de sete técnicas, consideradas as mais utilizadas no desenvolvimento de sistemas de apoio ao diagnóstico clínico. Outro processo necessário para avaliar a qualidade de análise foi segmentar a imagem em regiões de interesse, também seguindo outras sete principais técnicas, para verificar se a capacidade de diagnóstico e classificação da entropia amostral não era fortemente afetada. Por fim, um último teste a que os pesquisadores submeteram a análise computacional diz respeito ao nível de ruído das imagens. “Dentro da prática clínica, existem problemas que podem ser caracterizados como ruídos e que podem afetar a análise das imagens. Nós realizamos testes gerando de 20% até 80% de degradação nas imagens para verificar até que ponto era possível realizar a classificação correta dos linfomas”, explica Neves.
Para a pesquisa, o grupo utilizou 30 amostras histológicas, 10 para cada tipo de linfoma, e, a partir destas, foram geradas 375 imagens: 113 de Leucemia Linfoide Crônica, 140 de Linfoma Folicular e 122 de Linfoma de Células do Manto. Como resultado, o grupo foi capaz de identificar quais técnicas de tingimento e segmentação, associadas a um determinado nível de ruído, apresentaram resultados mais precisos quando combinadas à análise da entropia amostral, alcançando taxas de acerto entre 98,7% e 99,6%.
O conjunto de métodos encontrado pelos pesquisadores faz parte de um pacote que Neves espera que venha a ser utilizado como ferramenta de auxílio a especialistas em clínicas e hospitais tanto para o diagnóstico quanto para a classificação dos linfomas não-Hodgkin. Em artigo anterior, Sample Entropy Signatures: A new way to interpret SampEn values, publicado na revista científica Software Impacts, o grupo já havia disponibilizado o código desenvolvido para a técnica de entropia amostral. Já a publicação mais recente apresenta, além do código, o conjunto de técnicas que melhor funcionam em conjunto. “Nós tornamos públicos os cálculos que usamos para construir o método. Isso abre a possibilidade de que outros grupos, ao redor do planeta, contribuam para o aprimoramento e para a melhoria dessas técnicas”, diz Neves. Ele ressalta que no momento a pesquisa segue em fase laboratorial, buscando compreender os limites da capacidade do uso combinado das técnicas tanto em relação ao linfoma não-Hodgkin como também para o diagnóstico de outras doenças, incluindo a covid-19 e o câncer de colo retal.
Crédito da imagens: Shamir, L., Orlov, N., Mark Eckley, D. et al., 2008.